Search Thermo Fisher Scientific
RT-qPCR 基本原理
起始材料:RNA
定量逆转录PCR(quantitative reverse transcription PCR, RT-qPCR)是应用于以RNA作为起始材料的PCR实验中的一种实验方法。在该方法中,总RNA或信使RNA(mRNA)首先通过逆转录酶转录成互补DNA(cDNA)。随后,以cDNA为模板进行qPCR反应。RT-qPCR已被用于多种分子生物学的应用中,其中包括基因表达分析、RNA干扰验证、微阵列验证、病原体检测、基因测试和疾病研究。
RT-qPCR的一步法与两步法
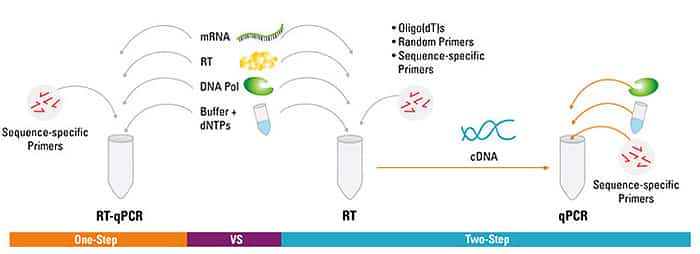
RT-qPCR可通过一步法或两步法来完成(图1、表1)。一步法RT-qPCR把逆转录与PCR扩增结合在一起,使逆转录酶与DNA聚合酶在同一管内同样缓冲液条件下完成反应。一步法RT-qPCR只需要利用序列特异性引物。在两步法RT-qPCR中,逆转录和PCR扩增过程是在两个管中完成,使用不同的优化的缓冲液、反应条件、以及引物设计策略。
图 1.一步法与两步法RT-qPCR
| 优点 | 缺点 | |
|---|---|---|
| 一步法 |
|
|
| 两步法 |
|
|
表 1.一步法及两步法RT-qPCR优缺点比较
总RNA与mRNA的选择
在设计RT-qPCR实验过程中,决定是否要使用总RNA或纯化的mRNA作为模板进行逆转录十分重要。尽管mRNA可能能够提供略高的灵敏度,但总RNA仍经常使用。其原因是总RNA作为起始材料具有较mRNA更重要的优势。首先,其过程需要较少的纯化步骤,这确保了更好的定量回收模板和更好的把结果标准化为起始的细胞数。其次,其避免了mRNA富集步骤,这能够避免由于不同mRNA的回收率不同而带来的结果偏移的可能性。总的来说,由于在大多数的应用中,目标基因的相对定量比检测的绝对灵敏度更为重要,因此在大多数情况下,总RNA更适用1。
逆转录引物
在两步法中,有三种不同的方法可用于引发cDNA反应:oligo(dT) 引物、随机引物、或序列特异性引物(图2,表2)。通常情况下,是将 oligo(dT) 引物和随机引物进行混合使用。这些引物退火至模板 mRNA 链,并提供给逆转录酶一个用于合成的起点位置。
| 引物选择 | 结构与功能 | 优点 | 缺点 |
|---|---|---|---|
| Oligo(dT) 引物(或锚定 oligo(dT) 引物) | 延展退火至 mRNA poly(A) 尾部的胸腺嘧啶残基;锚定 oligo(dT) 引物的3’端包含一个G、C或A (锚定位点) |
|
|
| 随机引物 | 长度为6至9个碱基,在 RNA 转录过程中,其可退火至多个位点 |
|
|
| 序列特异性引物 | 靶向特异的 mRNA 序列的定制引物 |
|
|
表 2.RT-qPCR 的 cDNA 合成中的引物注意事项。结合使用随机引物与锚定 oligo(dT) 引物可提高逆转录效率及 qPCR 的灵敏度。
逆转录酶
逆转录酶是利用 RNA 合成 DNA 的一种酶。一部分逆转录酶具有 RNA 酶活性,能够在转录后降解 RNA-DNA 杂交链中的 RNA 链。如果其不具有 Rnase 酶活性,可加入 RNaseH 以获得更高的 qPCR 效率。常用的酶包括莫洛尼鼠白血病病毒逆转录酶和禽成髓细胞瘤病毒逆转录酶。对于 RT-qPCR 来说,理想的情况下是选择具有较高热稳定性的逆转录酶,这样 cDNA 的合成能够在较高的温度下进行,确保成功转录具有较高二级结构的 RNA,同时保持其在整个反应过程中的全部活性,从而得到更高的 cDNA 产量。
逆转录酶的 RNase H 活性
RNase H 能够从 RNA-DNA 双链中降解 RNA 链,从而允许双链 DNA 的有效合成。然而,当使用长 mRNA 作为模板,RNA 可能被过早的降解,从而导致截短的 cDNA。因此,在 cDNA 克隆过程中,如果需要合成长的转录物时,尽量减小 RNase H 的活性通常是有益的。与此相反,拥有 RNase H 活性的逆转录酶通常有利于 qPCR 的应用,因为它们能够在 PCR 的第一个循环中提高 RNA-DNA 双链的熔解(图3)。
图 3.逆转录酶中 RNase H 活性。在 qPCR 中,使用具有 RNA 酶活性的逆转录酶。
如果 RNA 模板具有较高的二级结构建议操作该步骤。
该步骤建议用于引物的延伸。
逆转录酶以 mRNA 为模板合成 cDNA 链。
该步骤可阻止活性逆转录酶带来的 qPCR 抑制。
如果 RNA 模板具有较高的二级结构建议操作该步骤。
该步骤建议用于引物的延伸。
逆转录酶以 mRNA 为模板合成 cDNA 链。
该步骤可阻止活性逆转录酶带来的 qPCR 抑制。
RT-qPCR 的 qPCR 过程
引物设计
用于 RT-qPCR 中 qPCR 步骤的 PCR 引物最好应设计成跨越一个外显子-外显子连接,其中一条扩增引物可以潜在地跨越实际外显子-内含子边界(图4)。由于含内含子的基因组 DNA 序列不会被扩增,因此这种设计可以减少从污染的基因组DNA中扩增得到的假阳性的风险。
如果引物不能被设计成能够分离外显子或外显子-外显子边界,则有必要利用无 RNA 酶的 DNA 酶I或 dsDNA 酶处理 RNA 样品以除去基因组 DNA 污染。
RT-qPCR 对照
一个逆转录阴性对照(-RT对照)应该包括在所有的 RT-qPCR 的实验中,以检测 DNA 污染(如基因组 DNA 或来自之前反应的 PCR 产物)。这一对照包含除逆转录酶之外的所有反应组分。由于该对照不会发生逆转录,因此如果观察到 PCR 扩增,则极有可能来自 DNA 的污染。
图 4.RT-qPCR 中 qPCR 步骤的引物设计。1)如果一个引物被设计为跨越外显子-内含子边界,则可能造成污染的基因组 DNA 将不会被扩增,其原因是引物不能退火至该基因组 DNA 模板。相反,cDNA 不含任何内含子,能够有效被引物识别并扩增。2)当引物侧接一个长的内含子(例如1 kb),扩增反应将不会发生,因为短的延伸时间仅够用于扩增短的 cDNA,却不足以扩增基因组靶基因。
参考文献
- Bustin S. (ed) (2004) A-Z of Quantitative PCR.IUL Biotechnology Series, International University Line, La Jolla, California.
仅供科研使用,不可用于诊断目的。